Лаборатория молекулярной биоинженерии
|
Отдел молекулярной биологии и биотехнологии растений Руководитель: Мирошников Константин Анатольевич |
Лаборатория, занимающаяся комплексным молекулярно-биологическим исследованием вирусов бактерий (бактериофагов) была основана профессором Вадимом Викторовичем Месянжиновым в 1985 году в Институте вирусологии им. Д.И. Ивановского АМН СССР. В 1994-1997 гг. лаборатория базировалась в Институте биохимии им. А.Н. Баха РАН, с 1997 года входит в состав коллектива ИБХ РАН. С 2013 года лабораторию возглавляет Константин Анатольевич Мирошников.
Бактериофаги – наиболее многочисленные существа в природе, их разнообразие оценивается в 1015 таксономических видов. Многие процессы, составляющие ныне классику молекулярной биологии, были детально изучены на бактериофагах в качестве объектов исследования. Возможности лаборатории позволяют проведение полного цикла работ в области генной инженерии: от выбора стратегии клонирования индивидуального гена и его химико-ферментативного синтеза до разработки метода выделения, очистки, полной физико-химической идентификации и исследования биологической активности, масштабирования экспрессии и стадии ферментации. В лаборатории разработан алгоритм исследования бактериофагов с привлечением современных методов геномики, структурной и функциональной протеомики и физико-химических методов определения структуры белков и белковых комплексов.
1. Геномика бактериофагов
С 1999 года лаборатория участвует в международной программе исследования бактериофагов Pseudomonas aeruginosa.
Pseudomonas aeruginosa (синегнойная палочка) – чрезвычайно распространенная в почве и пресноводных водоемах бактерия, важная компонента в экосистемах. Бактериофаги, регулирующие баланс псевдомонадных популяций, соответственно, играют важную экологическую роль. Псевдомонады – условный патоген, способный вызывать внутрибольничные инфекции. Причём в силу природной и приобретенной антибиотикоустойчивости P.aeruginosa от этих инфекций достаточно сложно избавиться. Поэтому бактериофаги псевдомонад часто рассматриваются как дополнительный, и даже альтернативный метод терапии синегнойных инфекций. В настоящее время известно около 20 таксономических родов бактериофагов, инфицирующих псевдомонады, и их чисто постоянно растет. В зависимости от условий обитания штаммовое разнообразие P. aeruginosa весьма велико, соответственно, видовое разнообразие инфицирующих эти штаммы бактериофагов также открывает большие возможности для исследований. P. aeruginosa служит одним из модельных объектов исследования микробиома озера Байкал. Наша лаборатория участвовала в описании и утверждении родов phiKZ-подобных, KMV-подобных, PB1-подобных, М6-подобных бактериофагов Pseudomonas.
В настоящее время лаборатория ведет крупный проект по изучению и систематизации бактериофагов, инфицирующих сельскохозяйственные патогены. Так, энтеробактерии родов Pectobacterium и Dickeya вызывают черную ножку и мягкогнилостные бактериозы картофеля, которые приносят колоссальный урон картофелеводству. Для развития концепции биологической защиты урожая и контроля патогенов необходимо углубленное понимание генетической организации соответствующих бактериофагов. Секвенирование геномов бактериофагов проводится в сотрудничестве с ЦКП «Геном» ИБХФМ СО РАН (Новосибирск), Лимнологическим институтом СО РАН (Иркутск), Балтийским федеральным университетом им. И. Канта (Калининград), Katholieke Universiteit Leuven (Бельгия).
2. Протеомика бактериофагов. Функциональные белки (рецепторы, ферменты лизиса, шапероны)
Вирусы – мельчайшие представители живой природы, они отличаются небольшим и компактно организованным геномом. Однако до сих пор точная функция не менее трети генов вирусов до сих пор неизвестна. Выявление роли белков и пептидов, кодируемых генами бактериофагов – предмет интереса нашей лаборатории.
Основными объектами протеомных исследований служат ключевые структурные и функциональные белки, осуществляющие инфекционный цикл бактериофага:
- Рецепторные хвостовые белки. Инфекционный цикл бактериофага начинается со специфического присоединения вируса к поверхности бактерии и активного внедрения вирусной ДНК в цитоплазму. Присоединение осуществляется за счет белков хвостовых шипов и фибрилл бактериофага. Белки хвостовых шипов имеют очень прочные тримерные структуры с оригинальными белковыми фолдами и часто обладают дополнительной ферментативной активностью для разрушения защитных экзополисахаридов бактерий. Изучение ферментативных механизмов, субстратной специфичности и структуры таких белков имеет важное значение как для фундаментального понимания биологии бактериофагов, так и для прикладного применения рекомбинантных белков в белковой инженерии и для борьбы с бактериальными биопленками. Исследования проводятся в сотрудничестве с Институтом органической химии им. Зелинского РАН, Институтом микробиологии им. Виноградского РАН, Институтом биохимии им. Баха РАН, University of Texas Galveston (США), Universidade de Santiago de Compostela (Испания).
- Цитолитические ферменты (эндолизины). На завершающем этапе инфекционного цикла ферменты бактериофага разрушают хозяйскую клетку, облегчая выход дочернего поколения вирусов. Такие ферменты очень разнообразны по своим свойствам, структуре и субстратной специфичности. Рекомбинантные эндолизины нередко рассматриваются как «энзибиотики», противомикробные препараты для лечения антибиотикоустойчивых разновидностей патогенных бактерий. Исследования проводятся в сотрудничестве с Химическим и Биологическим факультетами МГУ, Институтом кристаллографии РАН, University of Texas ElPaso, Purdue University (США), Katholieke Universiteit Leuven (Бельгия).
- Молекулярные шапероны. Одна из важнейших проблем рекомбинантных технологий – сложности с получением целевого белка с природной пространственной структурой. Сворачивание полипептидной цепи белка в биологически активную форму (фолдинг) – часто неспонтанный процесс, который требует участия дополнительных факторов. Белки - ассистенты фолдинга, по первому описанному представителю этого класса, называют шаперонами. Хотя бактериофаги, как и все вирусы, зависимы от метаболических систем клетки-хозяина, в ряде фаговых геномов были обнаружены гены, кодирующие шапероны. Вероятно, эти гены были получены бактериофагами в результате горизонтального переноса и приспособлены для своих нужд. Исследование таких белков полезно для понимания механизмов фолдинга, который часто называют «третьей составляющей генетического кода». Также у белков бактериофагов часто встречаются «внутренние шапероны» – энергетически избыточные домены. Помимо быcтрого и спонтанного сворачивания в целевую структуру, такие домены («фолдоны» направляют сворачивание в нужную форму, присоединенную к ним часть полипептидной цепи. Использование шаперонов и фолдонов – привлекательный инструмент белковой инженерии. С его помощью был получен и исследован ряд белков, ранее неспособным к формированию активной структуры при синтезе в бактериальной клетке. Исследования проводятся в сотрудничестве с биологическим факультетом МГУ, Институтом кристаллографии РАН, University of Texas ElPaso (США).
3. Практическое применение бактериофагов (медицина, сельское хозяйство)
Россия – одна из немногих стран в мире, где препараты бактериофагов выпускаются промышленно и применяются в практической медицине в качестве антимикробных препаратов. Однако стандарты производства были разработаны более 30 лет назад, и перед изготовителями стоят задачи приведения препаратов в соответствие с современными требованиями, сформулированными научным и медицинским сообществами. Лаборатория молекулярной биоинженерии взаимодействует в разработке составов фаговых коктейлей и технологии производства и контроля с производителем медицинских фагопрепаратов ФГУП НПО «Микроген».
Также привлекательной стратегией представляется применение бактериофагов в сельском хозяйстве для борьбы с бактериозами растений. Взаимодействие в областях диагностики патогенов, подбора бактериофагов и производства препарата сельскохозяйственного назначения происходит с крупным научно-производственным агрохолдингом «Агропарк Рогачёво» (Московская обл.)
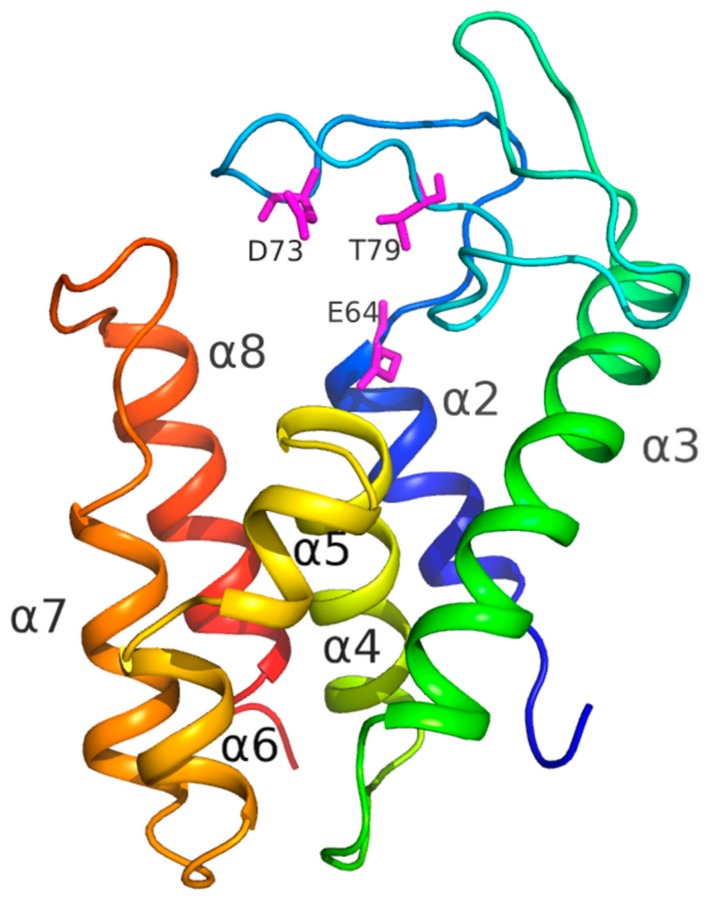 Ферменты, которые разрушают клеточную стенку бактерий, в свете возрастающей антибиотикоустойчивости патогенных микроорганизмов, считаются перспективными антибактериальными препаратами. Белки, гидролизующие пептидогликан (основной материал клеточной стенки), достаточно распространены в природе и кодируются в геномах многих бактерий (аутолизины) и бактериофагов (эндолизины). Основное препятствие для внедрения таких белков в практику – неспособность проникать через внешнюю мембрану грамотрицательных бактерий. Для осуществления этой функции либо используются дополнительные вещества, разрыхляющие мембрану, либо в полипептидную цепь фермента путем генной инженерии добавляется трансмембранный домен, обеспечивающий транспорт полученного химерного белка через мембрану бактерии. Такие белки называются «Артилизины» (ARTIficial LYSIN – «искусственный фермент»). При анализе бактериальных геномов нами был обнаружен фермент с предсказанной пептидогликан-гидролазной функцией, у которого на С-конце имеется последовательность, образующая положительно заряженную α-спираль с возможной трансмембранной функцией. Полученный рекомбинантный белок (AcLys) способен проявлять бактериолитическую активность на широком спектре грамотрицательных бактерий без добавления разрыхлителей мембран. Кристаллы белка AcLys были получены в условиях микрогравитации. Пространственная структура белка, полученная методом рентгеновской кристаллографии с разрешением 1.2 Å, показала наличие α-спирали (α8), не нарушающей целостность белковой глобулы и, вероятно, играющей активную роль в «лизисе снаружи». Исследование таких «природных артилизинов» способствуют рациональному дизайну «ферментативных антибиотиков».
Ферменты, которые разрушают клеточную стенку бактерий, в свете возрастающей антибиотикоустойчивости патогенных микроорганизмов, считаются перспективными антибактериальными препаратами. Белки, гидролизующие пептидогликан (основной материал клеточной стенки), достаточно распространены в природе и кодируются в геномах многих бактерий (аутолизины) и бактериофагов (эндолизины). Основное препятствие для внедрения таких белков в практику – неспособность проникать через внешнюю мембрану грамотрицательных бактерий. Для осуществления этой функции либо используются дополнительные вещества, разрыхляющие мембрану, либо в полипептидную цепь фермента путем генной инженерии добавляется трансмембранный домен, обеспечивающий транспорт полученного химерного белка через мембрану бактерии. Такие белки называются «Артилизины» (ARTIficial LYSIN – «искусственный фермент»). При анализе бактериальных геномов нами был обнаружен фермент с предсказанной пептидогликан-гидролазной функцией, у которого на С-конце имеется последовательность, образующая положительно заряженную α-спираль с возможной трансмембранной функцией. Полученный рекомбинантный белок (AcLys) способен проявлять бактериолитическую активность на широком спектре грамотрицательных бактерий без добавления разрыхлителей мембран. Кристаллы белка AcLys были получены в условиях микрогравитации. Пространственная структура белка, полученная методом рентгеновской кристаллографии с разрешением 1.2 Å, показала наличие α-спирали (α8), не нарушающей целостность белковой глобулы и, вероятно, играющей активную роль в «лизисе снаружи». Исследование таких «природных артилизинов» способствуют рациональному дизайну «ферментативных антибиотиков».
Sykilinda N.N., Nikolaeva A.Y., Shneider M.M., Mishkin D.V., Patutin A.A., Popov V.O., Boyko K.M., Klyachko N.L., Miroshnikov K.A.”Structure of an Acinetobacter Broad-Range Prophage Endolysin Reveals a C-Terminal α-Helix with the Proposed Role in Activity against Live Bacterial Cells” Viruses. (2018). V. 10. №6. pii: E309. doi: 10.3390/v10060309.
| ФИО | Должность | Контакты |
|---|---|---|
| Мирошников Константин Анатольевич, Чл.-корр. РАН, д.х.н. | г.н.с. | kmi@ibch.ru |
| Евсеев Петр Владимирович | с.н.с. | |
| Сыкилинда Н.Н., к.б.н. | с.н.с. | |
| Шнейдер Михаил Маркович, к.б.н. | с.н.с. | |
| Егоров Михаил Владимирович | м.н.с. | |
| Комаревцев Сергей Константинович | м.н.с. | |
| Тимошина Ольга Юрьевна, к.б.н. | м.н.с. | |
| Токмакова Анна Дмитриевна | м.н.с. | |
| Асеведо Солис П. | тех.-лаб. | |
| Головин Д.Л. | тех.-лаб. | |
| Ландышев Н.Н. | тех.-лаб. | |
| Путилов И.А. | тех.-лаб. | |
| Ткаченко Н.А. | тех.-лаб. | |
| Якимов А.Ю. | тех.-лаб. | |
Ранее здесь работали | ||
| Игнатов Александр Николаевич, д.б.н. | ||
| Воронина М.В. | ||
| Дорофеева Л.В. | ||
| Курочкина Л.П., к.х.н. | ||
| Кабанова А.П. | ||
| Лукьянова Анна Александровна, к.б.н. | a.al.lukianova@gmail.com | |
| Мирошников К.К. | ||
| Седов А.С. | ||
| Тиньков И.А. | ||
| Горносталь Е.А. | ||
| Дворякова Е.А. | ||
| Касимова А.А. | ||
| Пилик Р.И. | ||
| Рассказова П.М. | ||
| Тараканов Р.И. | ||
| Ширшиков Фёдор Владимирович | ||
| Месянжинов В.В., д.б.н. | ||
| Чертков О.В. | ||
 Загрузка...
Загрузка...Научные проекты
 Загрузка...
Загрузка... Загрузка...
Загрузка...
