Пресс-центр / новости / Наука /
Контекст стартового кодона взаимосвязан с наличием альтернативных стартов трансляции в основной рамке считывания
Сотрудникам ИБХ РАН вместе с зарубежными коллегами удалось обнаружить ранее неизвестную эволюционную зависимость между эффективностью стартовых кодонов и встречаемостью AUG кодонов в 5'-концевой части белок-кодирующих последовательностей. В том случае, когда старт кодоны мало эффективны, вероятность встречаемости следующего АUG кодона выше в той же рамке считывания. В то время как для высоко-эффективных кодонов встречаемость следующего АUG кодона выше в альтернативных рамках. Им также удалось показать что слабая инициация на первом старт кодоне связана с синтезом укороченных протеоформ обусловленных инициацией на втором старте.
Согласно современным представлениям о инициации белкового синтеза у эукариот, малая субьединица рибосомы с факторами инициации связывается с 5’концом мРНК, и затем начинает сканировать мРНК в поисках инициаторного кодона. После обнаружения стартового кодона (в большинстве случаев это AUG) происходит цепь событий, приводящая к диссоциации инициаторных факторов, к присоединению большой субьединицы рибосомы, и к началу синтеза полипептида. Эффективность узнавания стартового кодона зависит от его ближайшего нуклеотидного контекста. В 1980 годах Мэрилин Козак показала, что я эффективность контекста зависит в первую очередь от нуклеотидов в положении -3 и +4 (по отношению к нуклеотиду A кодона AUG). Оптимальный для инициации нуклеотидный контекст обычно называется контекстом Козак. Если же контекст не является оптимальным, то сканирующая рибосома может пропустить его и продолжить сканирование дальше – этот процесс известен как проскальзывающее сканирование (leaky scanning).
Сотрудники ИБХ РАН вместе с коллегами из МГУ, Университета Гранады (Испания) и Университетского Колледжа Корка (Ирландия) исследовали проскальзывающее сканирование на полногеномном и эволюционном уровне. Если в мРНК присутствует неоптимальный стартовый кодон, открывающий рамку считывания, в случае его “проскальзывания” возможны два варианта. Если следующий стартовый кодон расположен в той же рамке считывания, то в результате его узнавания рибосома будет синтезировать укороченный вариант белка. Если же следующий стартовый кодон находится в двух других рамках считывания, то будет синтезироваться совершенно другой полипептид. Маловероятно, что такой продукт инициации может иметь какую-то функциональную роль в клетке.
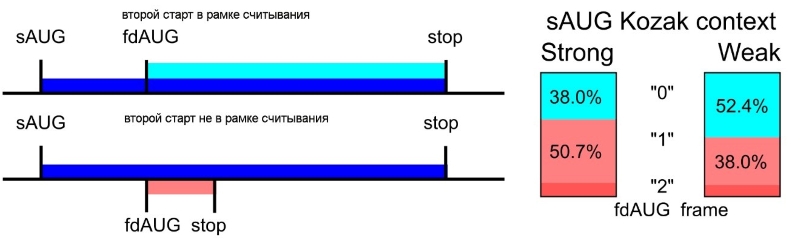
Слева: Схема двух конфигуарации встречаемсоти первых двух AUG кодонов. Справа: частота обнаружения таких конфигураций в зависмости от силы контекста первого старт кодона.
Исследуя взаимное расположение первого (основного) и последующего AUG кодона в мРНК, исследователи пришли к выводу, что частота нахождения второго кодона в рамке считывания зависит от контекста первого кодона. Чем хуже инициаторный контекст первого AUG кодона, тем чаще второй AUG кодон находится в той же, основной, рамке считывания. Более того, сравнивая консервативность контекстов стартовых кодонов мРНК у различных животных, был получен вывод, что низкая эффективность первого старта связана с высокой эффективностью второго старта (если он находится в той же рамке считывания), и что “сила” инициаторных кодонов в этих случаях более консервативна. Другими словами, архитектура мРНК, позволяющая синтезировать с одной мРНК два белковых продукта за счет проскальзывающего сканирования, сохраняется на эволюционном уровне для многих генов. Механизм проскальзывающего сканирования позволяет разнообразить протеом за счет синтеза укороченных с N-конца белков. Весьма вероятно, что такие измененные протеоформы могут иметь альтернативную внутриклеточную локализацию, или даже функциональную активность.
Работа опубликована в журнале Genome Research.
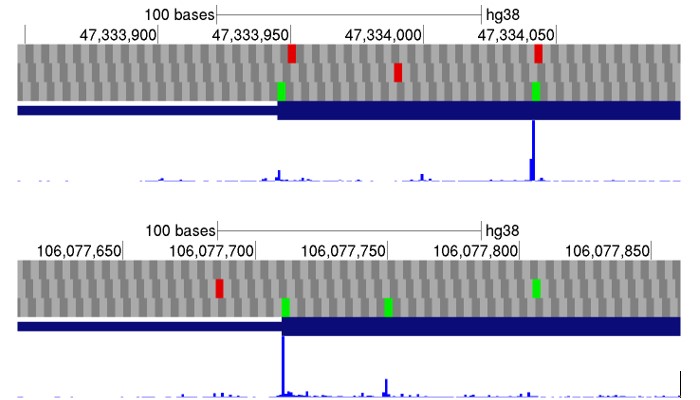
Примеры использования двух старт кодонов (AUG показаны зеленым) в человеческих генах подтверждаемые данными рибосомного профилирования (синие пики соответствуют рибосомам заблокированным ингибиторами инициации) из браузера GWIPS-viz (https://gwips.ucc.ie).
17 июля 2020 года




