Пресс-центр / новости / Наука /
Рождение виртуальной клеточной биологии
Системная биология обещает нам новые фундаментальные уровни понимания жизни, которых невозможно достичь без разработки количественных моделей клеток как целого и проверки сделанных ими предсказаний. В июльском выпуске журнала Cell Карр и соавторы представляют первую комплексную динамическую вычислительную модель бактерии, которая учитывает все ее компоненты и взаимодействия между ними.
Петр Р. Фреддолино(1) и Саид Тавазоие(1,*)
(1) Отдел биохимии, молекулярной биофизики и инициатив в системной биологии, Колумбийский университет, Нью-Йорк, США.
(*) Автор для переписки: st2744@columbia.edu
Научные дисциплины должны время от времени бросать вызов тем стандартам, которым соответствует их уровень понимания предмета. Безусловным стандартом в классической механике является способность предсказать динамическое поведение физической системы. Достигнет ли биология когда-либо такого же уровня понимания живой системы, пусть даже для отдельно взятой единичной клетки? Похоже, что именно сейчас настало время проверить наши знания о клетке, создав математическую модель всех клеточных процессов с использованием всех известных нам данных. Дьявол в этом проекте кроется в тысячах деталей, которые должны быть правильно учтены для того, чтобы в совокупности получить полную модель клетки как целого с приемлемой степенью точности. В этом выпуске (Karr et al. 2012)предлагают полную вычислительную модель бактерии Mycoplasma genitalium, которая учитывает влияние всех известных генов и их продуктов и позволяет провести моделирование полного клеточного цикла.
Предложенная авторами модель – это первая действительно всеобъемлющая попытка построить модель функционирования свободноживущего микроорганизма, которая достойна похвалы за свою исключительную смелость. Это гигантская задача, которая включает интерпретацию и объединение огромных объемов данных, многие из которых были взяты из других близких бактерий, таких как Mycoplasma pneumonia и Escherichia coli. Можно спорить о том, действительно ли доступная глубина и детальность наших знаний о работе отдельных элементов достаточна для того, чтобы в настоящий момент времени пробовать построить полную модель клетки. Бесспорно, многие скажут, что малочисленность и фрагментарность наших знаний, даже в отношении организма, геном которого близок к минимально возможному, заранее обрекает любую попытку подобного рода на поражение. Абсолютно точная, детальная, подробно разработанная математическая модель, безусловно, является мечтой отдаленного будущего. Тем не менее, в свете модели, предложенной (Karr et al. 2012), более уместным выглядит следующий вопрос: действительно ли мы уже знаем достаточно, чтобы попытаться провести грубое моделирование клетки и получить какие-либо нетривиальные результаты? Ответить на этот вопрос можно, только построив такую модель клетки, которая дает проверяемые предсказания. Это именно то, что сделали авторы, создав черновой набросок, к которому уже можно адресовать вопросы и получать ответы при помощи количественных моделей в масштабе целой клетки.
Модель предложенная (Karr et al. 2012)– это не первая попытка создать количественную, предсказательную, компьютерную модель масштаба целой клетки. Прежние попытки использовали модели, основанные на нелинейных дифференциальных уравнениях для моделирования биологических сетей (например, (Castellanos, Wilson, and Shuler 2004)) или моделирование, основанное на ограничениях, таких как анализ баланса реакций (FBA, flux balance analysis) и связанные с ними модели (Orth, Thiele, and Palsson 2010). Из всех предыдущих методов моделирования сетей клеточной регуляции и метаболизма, метод FBA заслуживает особого внимания в следствие широкого спектра успешных применений этого семейства методик; модели, основанные на ограничениях доказали свою пользу и качественное согласие с экспериментом в различных приложениях: от предсказания и объяснения эволюционных траекторий до метаболической инженерии (см. обзор (Feist and Palsson 2008)). Не смотря на многочисленные успехи, приведенные подходы, тем не менее, вынуждены ограничиваться некоторым подмножеством всех процессов клетки, поскольку до сих пор отсутствует методика расчета, применимая для моделирования всего разнообразия реакций и взаимодействий, происходящих в живой клетке. В дополнение к этому, метод моделирования, основанный на ограничениях, дает в качестве ответа стационарное состояние клетки, а вовсе не ее динамическую эволюцию, хотя в последнее время были сделаны определенные усилия чтобы включить FBA в моделирование динамических процессов (Min Lee et al. 2008).
Для того чтобы построить модель разнородных клеточных процессов, которые происходят в живых клетках на разных масштабах времени, (Karr et al. 2012)используют оригинальную модульную структуру данных, в которой многочисленные процессы живой клетки развиваются и взаимодействуют друг с другом. Модули, каждый из которых представляет собой отдельный класс процессов (например, транскрипция или метаболизм) разработаны, параметризованы и протестированы независимо друг от друга, в конечной модели они описывают вовлеченные в них компоненты на разных уровнях детализации. Модули взаимодействуют между собой и обмениваются переменными (которые все вместе описывают внутреннее состояние клетки) с интервалом в 1 сек; развитие этой модели во времени позволяет выполнить моделирование всего клеточного цикла M. genitalium. Модель оказывается весьма точной по ряду ключевых пунктов; авторы показывают, что их модель дает значения концентраций метаболитов, которые в пределах порядка величины совпадают с наблюдаемыми в живых клетках, кроме того, модель позволяет предсказать значимость (essentiality) генов M. genitalium с точностью 80%. Не менее впечатляет то, что модель дает существенно оригинальную гипотезу регуляции длительности клеточного цикла Mycoplasma: репликация генома в конечном счете лимитируется синтезом дезоксирибонуклеотид трифосфатов (dNTP), в результате клетки, в которых ранние стадии клеточного цикла заняли больше времени, догоняют тех, кто запустил репликацию раньше, используя большую концентрацию накопленных dNTP во время репликации, уменьшая тем самым разброс общей длительности клеточного цикла внутри популяции.
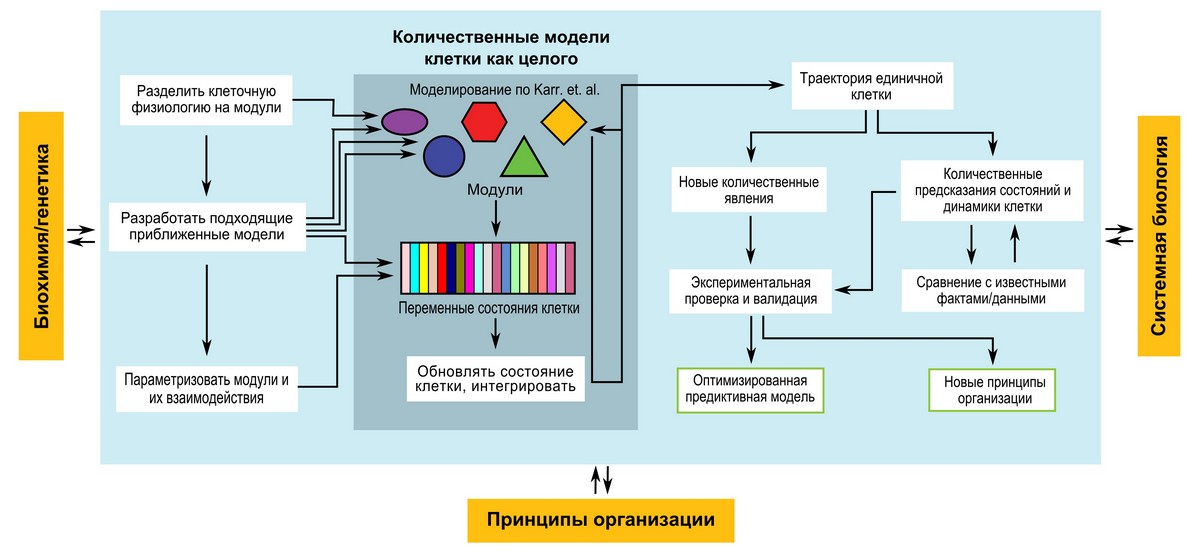
В свете представленного научного достижения важно ответить на два ключевых вопроса: (1) что мы ожидаем узнать из математической модели клетки как целого и (2) как такая модель должна взаимодействовать с другими областями биологии. Эти вопросы иллюстрирует рисунок 1, из которого видно, что современная биология находится на перекрестке трех направлений: качественные принципы, лежащие в основе организации живой материи; масштабные высокопроизводительные эксперименты, которые поставляют систематизированные количественные результаты и узкоспециальные модели конкретных систем и процессов клетки, полученные в результате специализированных исследований. Количественная модель клетки как целого обещает в перспективе принципиальную возможность объединить эти различные источники информации. В краткосрочной перспективе развитие и оптимизация подобных моделей сама по себе является серьезным вызовом; ученые, разрабатывающие эти модели, могут воспользоваться любым расхождением между их предсказаниями и известными экспериментальными фактами для того, чтобы уточнить структуру и содержание своих моделей. Мы можем себе представить два основных типа предсказаний, которые могут быть практически полезны: (1) открытие новых принципов организации, которые помогут собрать воедино наши знания о биологических системах («физическая перспектива») и (2) развитие достаточно точных вычислительных моделей, которые способны заменить, по крайней мере, предварительные стадии скринирования веществ в биоинженерии («инженерная перспектива»). Уникальный потенциал, который кроется в моделировании всех систем клетки – это способность проникнуть в детали процессов, которые невозможно изучить экспериментально. Действительно, такое моделирование может предоставить исследователю неограниченно подробные траектории развития единичных клеток, включая все параметры их внутреннего состояния, которые можно будет легко изменить для того, чтобы изучить любой интересующий нас феномен.
Чрезвычайно сложная, масштабная модель, представленная в работе (Karr et al. 2012)– это ключевой шаг на пути создания практичных и надежных методов моделирования клетки. Особенно впечатляет то, что эта изощренная модель дает нам не только приблизительное количественное согласие с разнообразными экспериментально измеренными параметрами, но и новое понимание механизмов регуляции биологических процессов. Тем не менее, мы должны особо подчеркнуть, что эта модель далека от идеальной «платонической» модели M. genitalium. Для каждого модуля, предложенного авторами модели, найдется эксперт, который выскажет справедливую критику в адрес математического описания модуля или оценки параметров модели, не смотря на то, что на настоящий момент они представляют собой наилучшую попытку достичь баланса между реализмом, вычислительной сложностью и количеством свободных параметров. Как обращают внимание сами авторы, данная модель представляет собой первый черновой набросок, более важный в качестве отправной точки для дальнейшего уточнения, чем модель, полезная сама по себе. В дальнейшем исследователям предстоит критически изучить все ошибки данной модели и определить, какие изменения структуры и параметров требуется внести для их устранения; по мнению авторов, чрезвычайно желательно расширение модели на экспериментально более изученные организмы, такие как E.coli. В дополнение к этому, для построения законченной реконструкции клетки разработчики моделей должны верифицировать те упрощения в организации клетки, которые пока еще не были рассмотрены: такие как наличие широко распространенных антисмысловых транскрипций в геноме (Dornenburg et al. 2010), эффекты пространственной гетерогенности (Roberts et al. 2011)и многофункциональность ферментов (Khersonsky and Tawfik 2010). Так же пока еще не вполне ясно, насколько уязвимы такие масштабные клеточные модели по отношению к неопределенностям в значениях параметров, которая характерна для моделей системной биологии (Gutenkunst et al. 2007), насколько хорошо общее поведение клетки будет предсказываться исходя из параметров, полученных в различных моделях и до какой степени можно будет полагаться на индивидуальные переменные состояния клетки даже если общая картина клетки как целого будет выглядеть корректной.
Перевод с английского: Максим А. Дубинный
Литература.
14 августа 2012 года
Источник: http://dx.doi.org/10.1016/j.cell.2012.07.001